Plant Physiol | 华中农业大学谢卡斌课题组利用RNA编辑技术揭示水稻OsDRB1-RNA的互作图谱
时间: 2023-03-20 点击次数:次 作者:
来源:
RNA结合蛋白(RNA binding protein, RBP)是一类重要的调节因子。RNA的加工、定位、周转、降解和功能行使等不同步骤都离不开RBP。RBP-RNA间的动态互作调控植物生长、发育、抗病和抗逆等不同生物学过程。在水稻中,双链RNA结合蛋白OsDRB1(double-stranded RNA binding proteins 1)不仅能够参与水稻中小RNA的产生与加工,并参与水稻对外界生物和非生物胁迫的响应。但在体内鉴定RBP的RNA配体一直是研究RBP功能的难点之一,新的RNA编辑技术为全基因组范围鉴定RBP-RNA互作提供了新策略。
近日,华中农业大学谢卡斌教授课题组在Plant Physiology在线发表了题为“Genome-wide profiling of OsDRB1-associated RNAs using targeted RNA editing in rice”的研究论文,使用RNA编辑技术详细地探究了OsDRB1互作的RNA配体。
该研究将一个腺苷脱氨酶ADAR的催化结构域(ADAR deaminase domain, ADARdd)与被研究的RBP进行融合表达(RBP-ADARdd),利用ADARdd对结合的RNA底物进行A-to-G编辑的能力,当RBP结合底物RNA后,融合蛋白中的ADARdd就可以将底物RNA进行A-to-G编辑。之后通过转录组测序并将测序结果与参考基因组进行比对,即可根据A-to-G(或T-to-C)的碱基变异位点识别出与该RBP结合的RNAs。
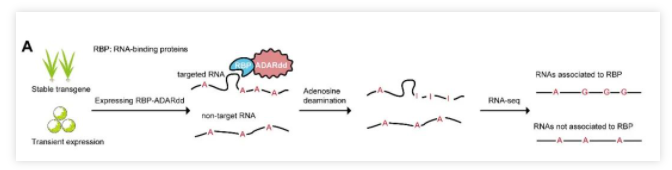
图1. RNA编辑鉴定RBP-RNA互作的示意图
根据该方法,课题组首先通过在水稻及拟南芥原生质体中分别利用MCP-MS2、AtGRP7的互作系统,验证了ADARdd在植物中的高效率RNA编辑能力,并发现RBP-ADARdd的编辑位点位于其结合位点41nt的范围之内。之后又通过遗传转化获得了超表达了OsDRB1-ADARdd的转基因水稻材料。经过转录组测序,与超表达OsDRB1或ADARdd的对照组水稻相比,在超表达OsDRB1-ADARdd的水稻根部及叶部样品中A-to-G以及T-to-C类型的碱基变异的数量显著高于其他几种类型的碱基变异,这些变异即为可能的由OsDRB1-ADARdd对OsDRB1结合的靶标RNA进行A-to-G的编辑造成的。课题组建立了一个严格的生信分析流程,可过滤掉99.7%-100%的基因组编辑位点、体细胞突变等背景,最终总共在根部和叶片的RNA样品中得到了1798个高度可信的编辑位点(High-Confidence Edits, HICEs)。
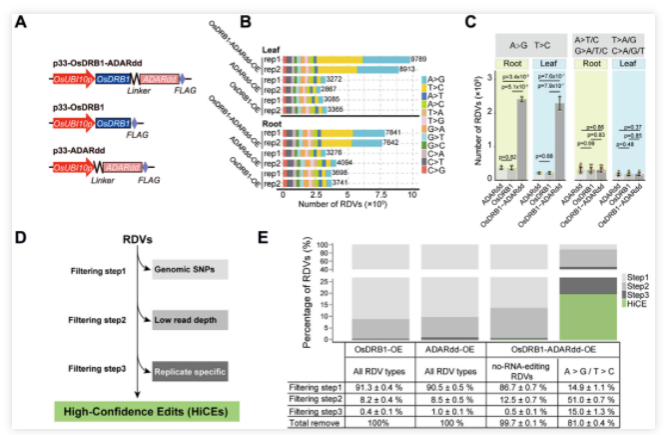
图2. OsDRB1-ADARdd编辑位点的鉴定
之后通过对这些HICEs位点进行分析,发现与OsDRB1互作的RNA具有一定的组织特异性。通过对HICE位点进行分析,发现OsDRB1主要结合非编码RNA以及mRNA的3’UTR以及内含子区域。其中,大部分的HICE位点主要位于水稻基因组上的重复区域,尤其是一些转座子区域,而这些区域恰好是水稻中小RNA产生的重要来源。之后课题组又通过小RNA测序(Small RNA-seq)并检测编辑位点,鉴定到18个已有注释的miRNA被编辑。最后课题组还将该方法的结果与传统研究RNA-RBP互作的RNA 免疫共沉淀(RNA Immunoprecipitation Sequencing, RIP-seq)方法进行了比较,证明通过位点编辑鉴定RBPs的RNA靶标是一种更加简便和高效的方法,为后续研究RBP-RNA的互作提供了方法和思路。
华中农业大学植物科技学院2019级博士研究生殷帅为该论文的第一作者,华中农业大学作物遗传改良全国重点实验室和洪山实验室谢卡斌教授为本文通讯作者,华中农业大学作物遗传改良全国重点实验室熊立仲教授也参与了该研究。该工作得到了国家自然科学基金、科技部转基因重大专项、华中农业大学自主创新基金等项目的资助完成。
论文链接:https://doi.org/10.1093/plphys/kiad158
